| Japanese top page / English top page |
PAICS
Parallelized ab initio Calculation System based on FMO
| プリオンタンパク質と低分子化合物に関してFMO計算を行った例を、これまでのプレゼンの資料を用いて紹介します。また、ここで紹介している研究内容は論文として出版されておりますので [1]、詳細はそちらを参照してください。 | |||
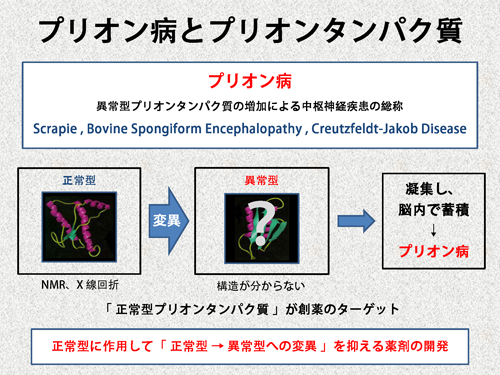
| プリオンタンパク質(PrP)は、スクレイピーやクロイツフェルト・ヤコブ病といったプリオン病の原因物質として知られており、正常型構造(PrPC)から異常型構造(PrPSC)への変異が病気を引き起こすと考えられています。そのため、PrPCに作用しPrPSCへの構造変異を抑える低分子化合物を発見することが、プリオン病の薬剤開発に繋がります。 | |||
 |
|||
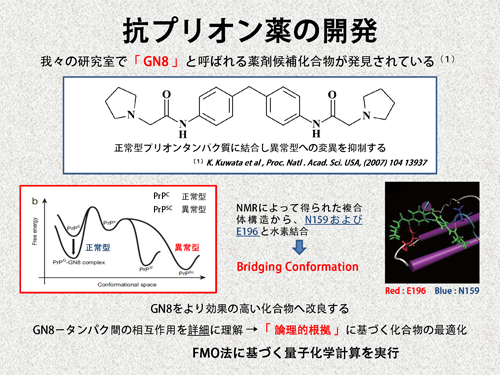
| 既に、我々の研究室では、GN8(下図)と呼ばれる薬剤候補化合物の発見に成功しています [2]。GN8は、PrPのN159とE196と水素結合を形成し、離れた2箇所を架橋することで、変異を抑制すると考えられています。しかし、薬剤としての利用を考えると、GN8を、さらに効果の高い化合物へ改良する(化合物の最適化)ことが望まれます。そこで、FMO計算によってPrPとGN8間の相互作用を詳細に解析し、化合物の最適化に役立つ情報を得ようと考えました。 | |||
 |
|||
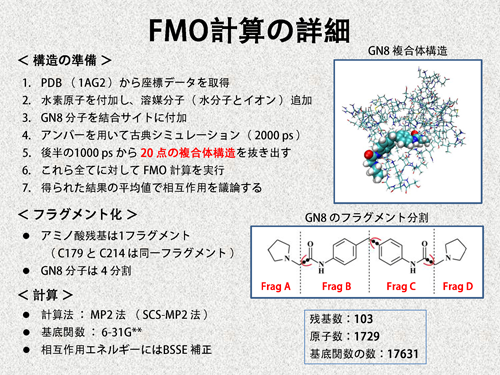
| 量子化学計算(FMO計算)を行うには、最初に、水素原子を含めた原子座標を準備しなければなりません。そこで、PDBの構造情報から複合体の初期構造を作成し、分子動力学計算を行いFMO計算に用いる原子座標を決めました。今回は、アミノ酸残基(103残基)を1つのフラグメントとし、GN8分子は4つのフラグメントに分割しました。また、計算には、van der Waals相互作用を考慮できるMP2法を用いました。 | |||
 |
|||
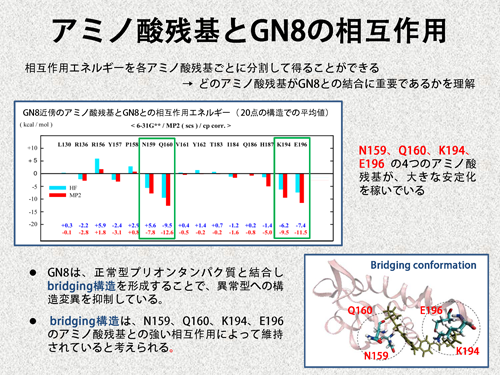
| FMO法では、フラグメント間の相互作用エネルギー(IFIE)が定義されます。よって、アミノ酸残基を1つのフラグメントとして計算した場合、残基ごとに分割した相互作用エネルギーが得られます。この情報から、リガンドとタンパク質の結合において、どのアミノ酸残基が重要であるかを知ることができます。GN8とPrPの場合、N159、Q160、K194、E196の4つのアミノ酸残基が、複合体の安定化に大きく寄与していました(下図)。つまり、構造変異の抑制に重要な架橋構造は、これらのアミノ酸残基との強い相互作用によって維持されていると考えられます。 | |||
 |
|||
| 今度は、GN8の4つのフラグメントに注目します。 先程と同様、フラグメント間の相互作用エネルギーから、GN8のどの部位がPrPとの結合に重要かを知ることができます。計算結果から、GN8の真ん中の部分(Frag BとFrag C)が、PrPと大きく相互作用していることが分かります。一方、GN8の両端の部分(Frag AとFrag D)はあまり相互作用しておらず、結合の安定化には寄与していません。このように、FMO計算を行えば、リガンドの各部位ごとに分割した相互作用エネルギーが得られます。 | |||
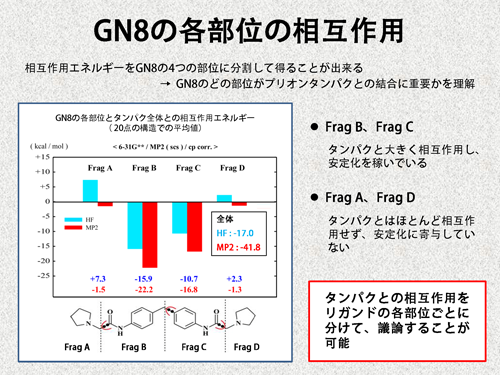 |
|||
| これらのデータが、創薬にどのように利用できるかを考えてみます。通常、量子化学計算で得られるのは、結合自由エネルギーのエンタルピー項(ΔH)への寄与です。よって、GN8のFrag BとFrag Cは、PrPとの結合において、ΔHを稼ぐ役割を果たしていると考えることができます。一方、Frag AとFrag Dは、ΔHにはほとんど寄与していません。よって、これらの部位は、結合自由エネルギーのエントロピー項への寄与や立体的効果を請け負っていると考えられます。現在、我々は、「リガンド分子の各部位の役割」を知ることで、化合物の最適化を効率的もしくは論理的に行う事を目指しています。 | |||
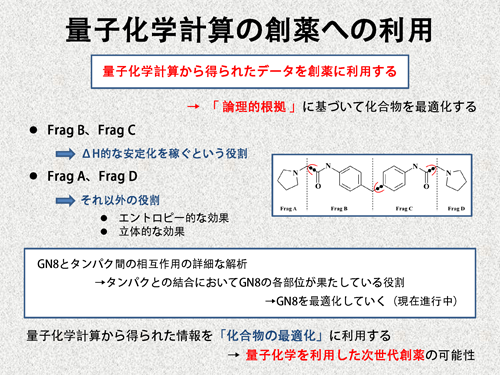 |
|||
|
ここで見たように、FMO計算を行うと、経験的ポテンシャルを用いずに、相互作用エネルギーをアミノ酸残基やリガンドの部位に分解して得ることができます。今回は、このような相互作用エネルギーの情報を創薬に利用することを考えましたが、創薬はFMO法を応用する可能性の一つに過ぎません。研究者のアイデア次第で、様々な分野に応用できると思います。
もし、ご自身の研究にFMO法を利用したいという方は、是非、PAICSを使ってみて下さい。また、ご自身の研究対象に関して、ここで示したようなデータを見てみたいという方がおられましたらご相談ください。場合によっては、共同研究という形をとり、FMO計算を我々が担当することも可能です。 |
|||
| [1] | Theoretical study of the prion protein based on the fragment molecular orbital method, T. Ishikawa, T. Ishikura, and K. Kuwata, J. Comput. Chem., 30 (2009) 2594-2601(DOI: 10.1002/jcc.21265) | ||
| [2] | Hot spots in prion protein for pathogenic conversion, K. Kuwata, N. Nishida, T. Matsumoto, Y. O. Kamatari, J. Hosokawa-Muto, K. Kodama, H. K. Nakamura, K. Kimura, M. Kawasaki, Y. Takakura, S. Shirabe, J. Takata, Y. Kataoka, and S. Katamine, Proc. Natl. Acad. Sci. USA, 104 (2007) 11921-11926 (DOI: 10.1073/pnas.0702671104) | ||
|